Résumé
- les variables ;
- les structures de contrôle ;
- les listes, les chaînes de caractères, les dictionnaires ;
- les fichiers ;
- les expressions rationnelles.
Objectif(s)
Compétences et apprentissages visés
- Enseignant: Benoist Gaston
- Enseignant: Thierry Lecroq

Volume d'enseignement
Cours : 10h
TP : 10h
Nombre de Crédits Européens
2
Modalités de Contrôle des Connaissances et de Compétences
Épreuve écrite CM : 20%
Épreuve écrite TP : 80%
Contenu de l'Unité d'Enseignement
- le langage de structuration de pages Hyper Text Markup Language HTML5
- le langage de description de style Cascading Style Sheet CSS3
- les extensions de type Canvas, SVG, MathML, Highcharts/Googlecharts etc
- les formulaires utilisateurs et les actions serveur (essentiellement en PHP)
- le langage à balisage markdown (utilisation du façonneur pandoc, de codimd, du markdown de github etc)
- une introduction au langage côté client JavaScript
Compétences et connaissances acquises à l'issue de cette Unité d'Enseignement
À l'issue de cet enseignement, l'étudiant pourra :
- comprendre les mécanismes à mettre en œuvre pour la conception d'un site www simple
- modifier et étendre un site www existant
- Enseignant: Laurent Mouchard

- Enseignant: Quentin Fouville
Cette UE majoritairement sous forme de TP, est consacrée à la prise en main pratique de l'analyse de données omiques. Elle comporte 2 matières. Elle est évaluée sous forme de projets.
- Analyse de donnés NGS 20h TP- elle s'appuie sur la partie théorique de l'UE Génomique Transcriptomique et les apprentissages de l'UE Programmation Python.

- Analyses de données en protéomique 25h. Elle s'appuie sur les apprentissages de l'UE Programmation Python.
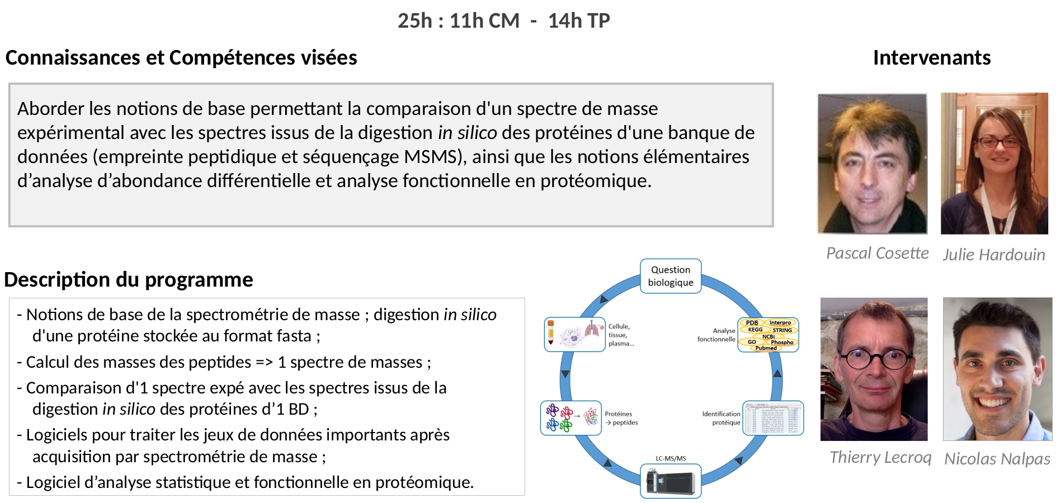
- Enseignant: Pascal Cosette
- Enseignant: Hélène Dauchel
- Enseignant: Julie Hardouin
- Enseignant: Thierry Lecroq
- Enseignant: Nicolas Nalpas
Programme: ce cours présente les approches et technologies de séquençage (NGS) et d'hybridation (Array) à très haut-débit qui permettent d'explorer les génomes, métagénomes, transcriptomes et régulomes. Quelque soit le domaine du vivant (microorganisme, végétal, animal, humain) ces technologies révolutionnent les connaissances fondamentales en biologie moléculaire (contenu des génomes, expression et régulation de l'expression des génomes) et trouvent des applications en agrosciences (agro-génomique), environnement (éco-génomique) pour les organismes et micro-organismes, en médecine génomique (génomique des cancers, des maladies complexes, médecine personnalisée, maladies infectieuses). Ce cours intègre la présentation des démarches et outils bioinformatiques et biostatistiques nécessaires au traitement des masses de données issues de ces grands projets d'exploration moléculaire des mécanismes du vivant.
Modalités : l'enseignement s'organise en CM, en TD (analyse de figures, lectures d'articles) et en TP en salle informatique (découverte des ressources internationales sur le web). Ce cours présente sa documentation essentiellement en langue anglaise. Il a lieu en présentiel 40h : 26h CM, 10h TD, 4h TP). L'UE est évaluée en contrôle continu intégral, par diverses travaux collectifs ou individuels; travaux dirigés, travaux pratiques et examen sur table. L'UE est obligatoire pour les deux parcours de la mention bionformatique (M1 BIMS, M2CCB4), pour le M1 mention Microbiologie et est optionnelle pour le M1 mention Agrosciences EcobioValo.
Pré-requis : programme de génétique moléculaire eucaryote et procaryote et génie génétique de niveau licence SV d'un parcours moléculaire. Cet UE ne comprend pas de programmation informatique, il n'y a aucun pré-requis dans cette discipline. Sur le plan des statistiques, ce cours fait appel aux connaissances en statistiques des tests.
A l'issue de ce cours les étudiants sont capables 1/ de se repérer dans le paysage général des approches haut débit et des applications en génomique, de comprendre leur évolution, potentiel et limitation 2/ de lire et comprendre dans des articles de diverses applications les objectifs, matériels et méthodes expérimentales et bioinformatiques/biostatistiques ainsi que les différentes figures de résultats des étapes (contrôle qualité, chaine de traitement, annotation, visualisation); 3/ d'identifier et d'utiliser les banques de données internationales du domaine et un Genome Browser. 4/ Les étudiants de la mention bioinformatique disposeront de l'ensemble des connaissances pré-requises pour aborder en pratique le traitement bioinformatique de données NGS (UE 3 analyse de données de séquençage et annotations).
- Enseignant: Hélène Dauchel

Volume d'enseignement
Cours : 8h
TP : 16h
Nombre de Crédits Européens
2
Modalités de Contrôle des Connaissances et de Compétences
Épreuve écrite TP : 100%
Contenu de l'Unité d'Enseignement
- environnement Linux et connexion à des serveurs distants avec ou sans VPN
- langage bash et son interface graphique yad
- aperçu des langages AWK et sed
- traitement de flux de données : présentation de certains pipelines (Nextflow, snakemake etc)
- machines virtuelles et containeurs : docker, singularity
Compétences et connaissances acquises à l'issue de cette Unité d'Enseignement
À l'issue de cet enseignement, l'étudiant pourra :
- créer ses propres scripts bash et modifier des scripts existants
- mettre en place une chaîne de traitement en utilisant un système de pipeline de type Nextflow ou snakemake ou autre
- utiliser docker de façon à travailler sur un container ou à créer un container
- Enseignant: Laurent Mouchard
Programme et objectifs:
Ce module a pour objectif la maîtrise des outils d'analyse, calcul et visualisation de données en Python: NumPy, SciPy, Pandas et Matplotlib. Le module permettra aussi une première prise en main de la bibliothèque Scikit-learn pour une initiation pratique à l'utilisation des méthodes d'apprentissage automatique. Le cours intègre, pour chaque outil, un apprentissage des techniques de manipulation et de traitement de données, il est ensuite mis en application lors de travaux pratiques. Les cas d'études traduisent des problématiques du monde de la bio-informatique comme la manipulation des données du génome humain de référence ou de la classification d'espèces. L'objectif étant de concevoir des projets qui vont de l’importation et le prétraitement de données nécessaires jusqu'à la visualisation des résultats de l'analyse.
Modalités d'évaluation:
L'enseignement s'organise en CM et en TP en salle informatique. Le cours présente sa documentation essentiellement en langue anglaise. Il a lieu en présentiel (18h : 6h CM, 12h TP). L'UE est évaluée en contrôle continu intégral par travaux pratiques.
Pré-requis:
Programmation Python.
Compétences attestées :
- Maîtriser les bibliothèques Python d’exploration de données et de calcul scientifique.
- Manipulation avancée de séries, de tableaux de données hétérogènes et de tableaux de calcul.
- Collecter des données, les organiser, les traiter, et les transformer en informations exploitables.
- Identifier les caractéristiques structurantes des données, les transformer et les visualiser.
- Programmer et résoudre un problème d’optimisation numérique (linéaire, non linéaire). Interpréter les résultats.
- Choisir et mettre en œuvre les différents modèles d'apprentissage. Mesurer, comparer et analyser la qualité des prédictions.
- Enseignant: Said Abdeddaim
Cette seconde UE entièrement pratique de programmation pour l'"analyse bioinformatique en sciences omiques" se compose de 2 parties :
- Web services et formats pour l'annotation (18h) : au cours de ces enseignements des outils pour l'accès programmatique aux ressources internationales (banques de données) sont étudiés (API Ensembl et API e-utilities du NCBI) ainsi que les formats usuels utilisés en annotation pour l'import-export de données dans des Genome Browsers. Un grand projet de développement de "Web services", transversal à cette matière et à d'autres UE (M1S2 Système de gestion de base de données, M1S1 Programmation 1) constitue l'évaluation : stratégies de scripts pour l’agrégation d’annotations des gènes et de leurs produits d’expression. Data Table (et Interface Web).
- Analyse de données en NGS (6h) ; après le Variant Calling et le RNA-seq au premier semestre, dans cette UE deux autres thématiques sont étudiées et s'appuie également sur la partie théorique de l'UE Génomique transcriptomique et l'UE de programmationn Python: un TP d'assemblage de génome complet (virus) est proposé (outils pour short reads et long reads) ; un TP d'analyse de données en métagénomique ciblée (metabarcoding 16S).
- Enseignant: Romain Coppee
- Enseignant: Hélène Dauchel
- Enseignant: Thierry Lecroq
- Enseignant: Arnaud Lefebvre
Cette seconde UE d'environnement professionnel est directement liées à la préparation du stage :
- Veille et Communication Scientifique (12h) : outils de recherche et veille avancée (MeSH PubMed), gestion bibliographique (format bibTeX, gestionnaire de références), rédaction de documents scientifiques (maitrise des styles et LaTeX, Beamer), canevas du rapport pré-stage.
- Enseignant: Hélène Dauchel
- Enseignant: Benoist Gaston
- Enseignant: Arnaud Lefebvre
- Enseignant: Elise Prieur-Gaston
- Enseignant: Fatima Soualmia-Dahamna
Cet enseignement se structure en deux thèmes abordés sous forme de cours, TD et TP en salle informatique :
- Phylogénie moléculaire : Origine de la phylogénie et concepts de bases en phylogénie moléculaire ; Caractéristiques des arbres (topologie, enracinement) ; Premières phylogénies moléculaires (horloge moléculaire, choix de l'ARNr, arbre du vivant). Règles de reconstruction des arbres : choix des séquences, alignement multiple de séquences et méthodes de reconstruction d'arbres phylogénétiques ; Estimation de la robustesse d'un arbre ; Applications et limites de la phylogénie. Application en virologie.
- Évolution des génomes : mécanismes moléculaires impliqués dans l'évolution de la structure et du contenu des génomes (duplications de génomes et de segments, duplication de gènes et fonction au sein des familles de protéines, implication des éléments mobiles, origine des introns); méthodes d’études (cartographie comparée, analyse de synténie, analyse comparative de séquences de génomes complets, outils et bases de données internationales de référence pour l'orthologie).
L'UE est évaluée en contrôle continu intégral par plusieurs devoirs écrits à la maison dans le cadre des TD ou TP (30%) et par deux devoirs sur table (70%), à part égale entre les deux parties . L'UE est obligatoire pour les parcours M1 BIMS et M1 MIC et est optionnelle pour le parcours M2CCB4. Pour le parcours BIMS ce cours est un pré-requis et sera réinvesti et approfondi sur les aspects algorithmiques et statistiques en M2 dans l'UE bioinformatique en génomique comparative. Ce cours s'appuie sur l'UE génomique ttranscriptomique pour sa partie connaissance des méthodes et procédures d'obtention de génomes complets, assemblage et annotation.
- Enseignant: Romain Coppee
- Enseignant: Hélène Dauchel
- Enseignant: Laurence Menu-Bouaouiche
Objectifs :
L'étudiant devra apprendre à maitriser la statistique bivariée tout d'abord en comprenant la notion de corrélation puis en s'appropriant les différentes étapes de la modélisation statistique, du choix du modèle à son utilisation en passant par la validation et la qualité de ce modèle.
Contenu :
_ Corrélation entre deux variables (Pearson, Spearman): notion et tests de corrélation.
_ Régression linéaire simple : les différentes étapes de la modélisation sur des exemples.
_ ANOVA : tests de comparaisons de plusieurs moyennes
_ Régression non-linéaire simple et problème de choix de modèles.
La totalité de l'enseignement se fera en salle machine en utilisant le logiciel R. L'enseignement se fera par des études de cas : présentation du problème biologique, du protocole expérimental et des données obtenues puis un traitement statistique.
- Enseignant: Caroline Berard
Évaluation : Devoir Maison + CC écrit
Programme :
• Statistique exploratoire multidimensionnelle : ACP, AFC.
• Classification supervisée : arbres de décision, k-plus proches voisins.
• Classification non supervisée : Kmeans, CAH, Modèles de mélange.
• Mise en œuvre sous R. Manipulation du logiciel R, fonctions graphiques pour l'exploration et l'analyse de données.
Pré-requis :
• savoir : bases d'algèbre linéaire, diagonalisation de matrices, changement de bases.
• savoir-faire : logiciel R.
• équivalent UE existante : M1S1 Algèbre linéaire.
- Enseignant: Caroline Berard
Série de quatre UE à chaque semestre du master. Elles sont dédiées à la préparation des démarches professionnelles.
- M1 S1 - Environnement professionnel 1 - Métiers de la Bioinformatique du M1S1 (10h TD + travail personnel; 1 CE) présente les domaines et les carrières en bioinformatique, accompagne la recherche du stage de S2 des M1BIMS/M2CCB4 et prépare dans le même temps à l'alternance en M2 BIMS : recherche d'un stage conventionné en alternance (formation initiale) ou d'un contrat d'apprentissage (apprenti).
- Enseignant: Hélène Dauchel
- Enseignant: Hélène Dauchel
- Enseignant: Caroline Berard
Programme : ce cours présente les enjeux de la médecine génomique personnalisée et le rôle essentiel de la bioinformatique dans ce contexte. En effet, depuis l’avènement du séquençage de nouvelle génération, le défi majeur en génétique médicale n’est plus l’identification des variations génétiques dans le génome des patients mais leur interprétation biologique et clinique. Les enseignements proposés dans le cadre de cette UE présentent les différents types de variations génétiques et leurs nomenclatures ainsi que les méthodes d’évaluation du caractère pathogène de ces variations (mutations constitutionnelles, altérations du nombre de copies, mutations somatiques et cancer). Ils intègrent la description de multiples approches expérimentales et bioinformatiques permettant d’appréhender l’impact fonctionnel des variations génétiques.
Modalités : cette UE s’organise en cours, en TD et en TP incluant la présentation de publications scientifiques récentes, de grandes ressources internationales et une sensibilisation aux questions éthiques en génomique. Elle est est évaluée en contrôle continu intégral, par deux travaux d'exposés (50%) et par un examen écrit sur table (50%). L'UE est obligatoire pour le parcours M1 BIMS et optionnelle pour le parcours M2CCB4.
Pré-requis : programme de génétique moléculaire eucaryote et procaryote et génie génétique de niveau licence SV d'un parcours moléculaire. Cette UE ne comprend pas de programmation informatique, il n'y a aucun pré-requis dans cette discipline.
A l'issue de ce cours les étudiants auront découvert l’importance des différentes bases de données mutationnelles et des grands projets internationaux de séquençage en génomique humaine. L’ensemble de ces enseignements doit aussi permettre aux étudiants d’acquérir des concepts génétiques et moléculaires fondamentaux dans le contexte de pathologies humaines. Au travers de cette UE combinée aux acquis de l'UE Génomique Transcriptomique, les étudiants disposeront de l'ensemble des connaissances pour aborder en pratique le traitement bioinformatique de données NGS en génomique des cancers (UE 3 Analyse de données de séquençage et annotations).
- Enseignant: Hélène Dauchel
- Enseignant: Pascaline Gaildrat
